OpenSSL
OpenSSL est une implémentation libre des algorithmes de chiffrement SSL basée sur un ensemble composé d'un outil en ligne de commande et de deux bibliothèques sous formes de librairies indépendantes :- Une librairie de cryptographie générale : libeay
- Une librairie implémentant le protocole SSL : SSLeay de Eric Young et Tim Hudson
Grâce aux wrappers, il est possible d'utiliser les bibliothèques OpenSSL dans une grande variété de langages de programmation.
Les paramètres de la commande en ligne OpenSSL sont très nombreux ; ils permettent d'indiquer entre autres l'un des nombreux types de chiffrement (exemple : blowfish, DES ou Triple DES, DSA, RC4, RC5, RSA...), d'encodage (base64 ou autres) et de hachage (MD5, SHA-1...). L'outil peut fonctionner en mode client et en mode génération de contenu.
Cet utilitaire et les bibliothèques associées sont disponibles pour la plupart des environnements et sont sous une licence de type Apache.
Les structures SSL BIO
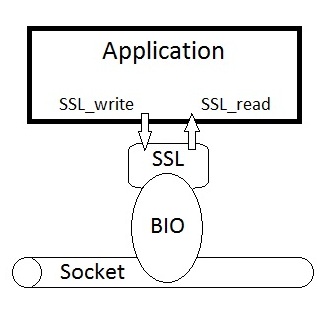
La couche BIO est une couche d'abstraction d'entrée/sortie, grâce à celle-ci on peut lire dans un fichier classique, comme communiquer via un socket protégé par SSL.Les chaînes
Cette couche contient deux types d'éléments, les sources (socket, fichier, ...) et les filtres (chiffrement, buffer, ...).Ces éléments sont reliés ensemble selon les besoins de l'utilisateur, en une double liste chaînée. Un exemple classique est la chaîne BIO utilisée par un client SSL :
Le premier élément de la chaîne est le premier à être appelé lors de toute opération de lecture/écriture. Par exemple, si l'on fait une opération read sur buffer, celui si va faire une opération read sur SSL, qui fera lui-même une opération read sur socket. Le dernier élément doit donc être une source, et les autres des filtres.
La structure des éléments
Un BIO_METHOD est un type, qui contient les opérations nécessaire au BIO :
typedef struct bio_method_st
{
int type;
const char *name;
int (*bwrite)(BIO *, const char *, int);
int (*bread)(BIO *, char *, int);
int (*bputs)(BIO *, const char *);
int (*bgets)(BIO *, char *, int);
long (*ctrl)(BIO *, int, long, void *);
int (*create)(BIO *);
int (*destroy)(BIO *);
long (*callback_ctrl)(BIO *, int, bio_info_cb *);
} BIO_METHOD;
Pour créer un nouvel élément, il nous faut récupérer le BIO_METHOD de notre élément.Pour chaque type de maillon BIO, il existe une fonction permettant de récupérer ce BIO_METHOD, son nom est formé comme suit :
// sources
BIO_METHOD *BIO_s_accept(void);
BIO_METHOD *BIO_s_connect(void);
BIO_METHOD *BIO_s_socket(void);
BIO_METHOD *BIO_s_fd(void);
// filtres
BIO_METHOD *BIO_f_ssl(void);
BIO_METHOD *BIO_f_buffer(void);
// sources et filtres divers en vrac
BIO_METHOD *BIO_s_mem(void);
BIO_METHOD *BIO_s_log(void);
BIO_METHOD *BIO_s_datagram(void);
BIO_METHOD *BIO_s_bio(void);
BIO_METHOD *BIO_s_null(void);
BIO_METHOD *BIO_f_null(void);
BIO_METHOD *BIO_f_linebuffer(void);
BIO_METHOD *BIO_f_nbio_test(void);
La structure d'un maillon est complexe, mais il y a peu à gérer pour créer un nouveau type de maillon :
typedef struct bio_st
{
BIO_METHOD *method;
long (*callback)(struct bio_st *,int,const char *,int, long,long);
char *cb_arg;
/* partie importante si on crée un nouveau type de maillon */
int init;
int shutdown;
int flags;
int retry_reason;
int num;
void *ptr; // on stocke nos données liées
struct bio_st *next_bio;
struct bio_st *prev_bio;
int references;
unsigned long num_read;
unsigned long num_write;
CRYPTO_EX_DATA ex_data;
} BIO;